�} 1 �I�C���[�O���t�ƗL���n�~���g���O���t
�@�R���s���[�^�Ƃ����Ύ������́A�ʏ�A�t�H���m�C�}���^�̂o�b�ʼnt����ʂ� ���āACPU������A�������[������A�n�[�h�f�B�X�N������A�L�[�{�[�h������ƁA ���݂ł́A������O�̂悤�Ɏg�p���Ă���v�Z�@��z������B�������A�w1936�N�� �p���̐��w�҃`���[�����O���A�v���O�����\�Ȕėp�v�Z�@�̊T�O���l�Ă����Ƃ��A �u�R���s���[�^�v�Ƃ������t���Ӗ�����̂́A�v�Z�@�ł͂Ȃ��A"�v�Z�����"�܂� �l�Ԃ��w���̂����ʂ������x�悤���B[6] �R���s���[�^�Ƃ����Ă��A���܂̂悤�ȃn �[�h�łȂ��Ă��ǂ��킯�ł���B�b�肪����邪�ȑO�A���͗c�����ɁA�N�ɂł��A�P �x�͍l����悤�ȏ����s�v�c�ȋ^��ɂ܂Â����B����́A�l�̎�͓�����O�̂悤 �ɓ����Ă��邪�A�ǂ��ŁA������𐧌䂵�Ă���̂��Ƃ������Ƃł���B�������A �]����w�߂��ł�킯�����A�]�ȊO�ŏ������߂Ă����ꏊ�A�����DNA�Ƃ������q�� �����Ȃ��Ă���BDNA�́A�זE���ł���ς��������邽�߂̕������̋@�\�Ƃ��� �������̏����q���̍זE�ւƎp�����邽�߂̎��ȕ����̋@�\�����������邽�� �ɗ��p����Ă���B��w���w�����A�R���s���[�^�Ƃ������̂ɂ��܂�ւ�������Ƃ� �Ȃ����͋��ȃv���O�����̎��Ƃ��Ȃ���˘f���������Ă����B���ƂȂ��ẮA�� ���̂��Ƃł͂��邪�A�v���O�����������Ă���notepad������v���O�����łł��Ă��� �̂ł͂Ȃ����ƁB�R���s���[�^�Ƃ����ē�����O�Ɏd���̂����Ƃ��đ��݂��Ă���@�B �̓G���W�j�A���g����̎g���₷���悤�ɈӐ}���Đ����������̂Ȃ̂ł���B�������� �����̂��Ƃ����ƁA�R���s���[�^�͍��A���݂���悤�Ȍ`�����Ă��Ȃ��Ă��悢�̂ł� �Ȃ����Ƃ������Ƃł���B
�@DNA���g���Čv�Z���s�����l���������BRSA�Í��̊J���҂̈�lLeonard M. Adleman�� �A1994�N��DNA���g���ANP���S���ł���L���n�~���g���p�X�����������B��������A DNA�R���s���[�^�Ƃ������삪���܂ꂽ�B
�@�{�_���́AAdleman �̘_����A Sticker-Based Model For DNA computation�̃��r���[ ���s���BAdleman�̘_����DNA �R���s���[�^�Ƃ������삪�ł������������ƂȂ�_���ł��� ������ł���B�܂��AA Sticker-Based Model For DNA Computation �̘_���́AAdleman �Ɠ���NP���S����DNA���g���ĉ������_����RAM�̊T�O���͂��߂�DNA�R���s���[�^�Ɏ� ����ꂽ�_���ł���B�܂��A�����Œ�Ă��ꂽstickers model���g���A�����ʼn��Z���s���R���s�� �[�^�V�X�e���̃A�C�f�A���Љ��Ă���B
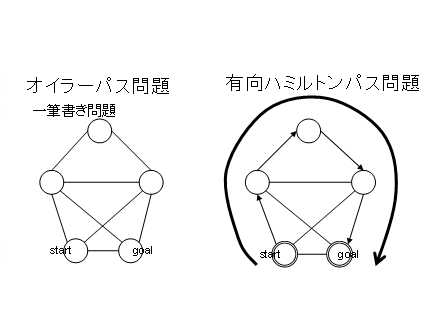
�@�O���t�Ƃ͓_�Ƃ��������Ԑ��̏W���̂��Ƃ������B�O���t�ƕ����ƁA�_�O���t�A�~�O�� �t�Ȃǂ������̐��l��}�\�Ŏ��������̂̂悤�Ɏv�����A����͈Ⴄ�B�_�̂��Ƃ_ �Ƃ����A���͕ӂƂ����B�O���t�̕ӂɌ������K�v�ŕӂ���ł͂Ȃ����ŕ\������ꍇ�� ����B���������O���t��L���O���t�Ƃ����B�אڂ��Ă��钸�_���m�����ǂ����ӂ̌n��� �����i���E�E�H�[�N�j�Ƃ����B�ӂ̏d���������Ȃ��ꍇ�A�H�i���a�E�g���C���j�Ƃ����A ���_�̏d���������Ȃ��ꍇ�A���i�p�X�i�J�����������p�X�Ƃ����ꍇ�͒P���p�X�j�j�Ƃ� ���B[7]

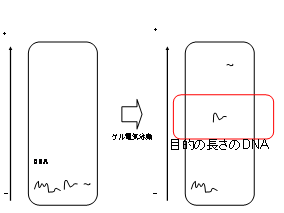
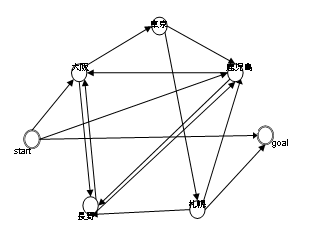
�}1.�̃O���t���l����B���̓I�C���[�O���t�B�E�͗L���n�~���g���O���t�Ƃ����O���t�� ����B�I�C���[�O���t�Ƃ͂��ׂĂ̕ӂ�1�x�����ʂ�H�����O���t�̂��ƂŁA�����I�ɂ͈� �M�������Ƃł���B�I�C���[�p�X���Ƃ͈�M�����\���ǂ�����₤���ł���B�n�~���g ���O���t�Ƃ́A����Ȓ��_start�������Ȓ��_goal�܂őS�Ă̒��_����x�����ʂ�p�X���� �݂��邩�A���Ȃ�����₤���ł���B�ꌩ�A�I�C���[�O���t�̕���������ɂ݂��邪�A�� ��̓���Ƃ��ẮA�n�~���g���O���t�̕�������̂ł���B�ł́A��̉������̓�� �����߂Ă���̂ł��낤���B
�@�ʏ�̃R���s���[�^�ő��������Ԃɉ��邱�Ƃ��ł�����̏W����P�Ƃ����B
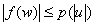
�����I�ɐ�������ƁA�@5,3,1,4,6,2�Ƃ����������͂��ꂽ�Ƃ��ɁA6���ő�l��
����Ƃ����̂́A���͒��ɔ�Ⴕ�����Ԃ�������B�܂��A
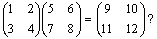 �Ƃ����s�������ǂ����肷��ɂ́A�s��̐ρAn�~n�Ȃ�
�Ƃ����s�������ǂ����肷��ɂ́A�s��̐ρAn�~n�Ȃ� ������B�������A�Ƃ��ɒʏ�
�̃R���s���[�^�ʼn���̂ɑ��������ԂŎ����\�ł���B���̂悤�Ȗ���P�̖��̏W��
�ɑ�����B
������B�������A�Ƃ��ɒʏ�
�̃R���s���[�^�ʼn���̂ɑ��������ԂŎ����\�ł���B���̂悤�Ȗ���P�̖��̏W��
�ɑ�����B
�@���������Ԃʼn����Ȃ��������݂���B��Ƃ���
�Ȃǂ����邪�A���̒ʏ�̃R���s���[�^�ł͑��������Ԃʼn����Ȃ��ł��낤���̏W���̒� ��NP�����݂���BNP�Ƃ́u���ɑ��đ��������Ԓ��̃q���g������AYes, No�𑽍����� �ԂŔ���ł�����v�ł���BP�͂���A�u�����̂��ȒP�Ȗ��v�̏W���ANP�́u�������� �����ȒP�Ȗ��v�̏W���Ƃ�����̂ł���
�@�}1�̃I�C���[�O���t����P�ɑ�����B�L���n�~���g���p�X����NP�ɑ�����B���̓��
�O���t�����ꂼ�ꑮ����W�������������ǂ����B�����P=NP���Ƃ����A���̖�肪����
��A�܋���100�����o�鐔�w��7����̈�ł���B���݂ł�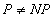 �Ɨ\�z����Ă���B�܂�
�ANP�͒ʏ�̃R���s���[�^�ł͑��������Ԃʼn����Ȃ��Ɨ\�z����Ă���̂ł���B
�Ɨ\�z����Ă���B�܂�
�ANP�͒ʏ�̃R���s���[�^�ł͑��������Ԃʼn����Ȃ��Ɨ\�z����Ă���̂ł���B
�@�}1.�̃I�C���[�O���t���͖�肪�����邩�ǂ����̃`�F�b�N���@�����݂���B����͒� �_�ɕӂ���{�Ȃ����Ă��钸�_����_�Ƃ����̂����A��_�̐���0��2�ł���A���̃I �C���[�O���t�͈�M�����\�ł���Ƃ킩��B�������M��������_�̌��ɋA���ł���� �����B �܂��A��M��������_�̌��ɋA���ł���Ƃ������Ƃ�
�ˁ@��M�����͊�_�̌��قǓ���Ȃ�
�ˁ@��M���� ��_�̌�
��_�̌�
�Ƃ����W������B
�@���A�����B�ɋA�������Ƃ��AB�̉�@��A���������߂Ɏg�����Ƃ��ł���B�v�Z�̌��� ���l���ɓ��ꂽ�A���\�����`����B���̂Ƃ��A���A�����B�Ɍ����I�ɋA���ł���Ȃ� �AB�ɑ�������I�ȉ�@��A�������I�ɉ������߂Ɏg�����Ƃ��ł���B
�C�ӂ̓���w�œ�����J�n���A�e�[�v��f(w)�����������o���Ē�~����悤�ȑ��������� Turing�@�BM�����݂���Ƃ��A��f :���� ������ �͑��������Ԍv�Z�\���ł���B
����A��B�ɑ��āA���ׂĂ�w�ɂ���
�@�@�@�@w��A��f�iw�j��B
�ł���悤�ȑ��������Ԍv�Z�\��f :���� ������ �����݂���Ƃ��AA B�Ə����A����A
�͌���B�ɑ��������ԋA���\�ł���Ƃ����B��f�́AA����B�֑��������ԋA���Ƃ���B
B�Ə����A����A
�͌���B�ɑ��������ԋA���\�ł���Ƃ����B��f�́AA����B�֑��������ԋA���Ƃ���B
�@A����B�ւ̑��������ԋA���́AA�̗v�f���ǂ����̔����B�̗v�f���ǂ����̔���ɕϊ��� ����@��^����B���������āA�A��f��p����Aw��A�ł��邩�肷�邽�߂ɂ́A f�iw�j��B�ł��邩�肷��悢�B�������Af�̌v�Z�͌����I�ɍs����B���̐������g ���ƁAB�̔�������������������ԃA���S���Y����p���āAA�̔�������������������� �A���S���Y�����\���ł���B[5] ���A�t��A���������Ƃ�����ł��邱�Ƃ��킩���Ă���ꍇ �Af�̑��݂ɂ��B���������Ƃ�����ł��邱�Ƃ��킩��B
�@1970�N�㏉����Stephen Cook ��Leonid Levin�́A�X�̖��̕��G����NP�̃N���X�S�̂� ���G���ƊW�Â�����悤�ȁA���̂悤�Ȗ��������B�����̖��̂�����ɂ��� ���đ��������ԃA���S���Y�������݂���Ȃ�ANP�ɑ����邷�ׂĂ̖��͑��������Ԃ� �������Ƃ��ł���̂ł���B���̂悤�ȓ���������NP����NP���S�ƌĂ��B[5]
���̏������݂�������B��NP���S(NP-complete)�Ƃ����B
 | i �Ԗڂ̔萫�`���[�����O�@�BNi ��x �������
| i �Ԗڂ̔萫�`���[�����O�@�BNi ��x �������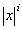 +i �X�e�b�v�ȓ���accept����}
+i �X�e�b�v�ȓ���accept����}
�@P=NP���̌����P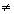 NP���\�z����Ă���B�܂�A�L���n�~���g���p�X���̓R���s���[�^
�ł͉����Ȃ����Ȃ̂ł���B�������A���݂ƌ����̈Ⴄ�R���s���[�^��p���邱�Ƃ�NP���S��
��ł���L���n�~���g���p�X�����������l��������B
NP���\�z����Ă���B�܂�A�L���n�~���g���p�X���̓R���s���[�^
�ł͉����Ȃ����Ȃ̂ł���B�������A���݂ƌ����̈Ⴄ�R���s���[�^��p���邱�Ƃ�NP���S��
��ł���L���n�~���g���p�X�����������l��������B
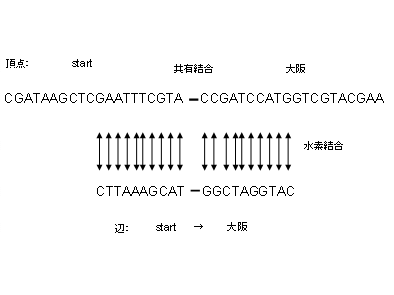
�@DNA�Ƃ�Deoxyribo Nucleic Acid �̗��Ńf�I�L�V���{�j�_�̂��Ƃł���BDNA�̓k�N���I�`�h�� ���ɘA�Ȃ����������ł���B���̉�������2��ɕ��т��݂������t���������ƂŐ}2�E�}�̂� ����2�d�点����`�����Ă���BDNA�͂���ς��������邽�߂̕������̋@�\�Ƃ��̕����� �̏��i��`���j���q���̍זE�ւƎp�����邽�߂̎��ȕ����̋@�\�̂��߂ɐ��̓��ŗ� �p����Ă��镪�q�ł���B
�@DNA���\�����Ă���k�N���I�`�h�͓��A�����_�A4��ނ̉����\������Ă���B4��ނ̉��� �̓A�f�j���A�O�A�j���A�V�g�V���A�`�~���ł���B4��ނ̉���͂��ꂼ��AA�AG�AC�AT�Ɨ��� ��B���ꂼ��̓��ƃ����_�͓����\�������Ă���̂ŁA�k�N���I�`�h��A�AG�AC�AT�ŕ\������ ���Ƃ��ł���B�܂�DNA��A�AG�AC�AT�ŕ\�����邱�Ƃ��ł���B�܂��A�d�v�Ȃ��ƂɁADNA�� �\������A�AG�AC�AT�����ŕ\�����邱�Ƃ��ł���̂ł���B�k�N���I�`�h�̘A�Ȃ������̂� DNA���ƕ\������B
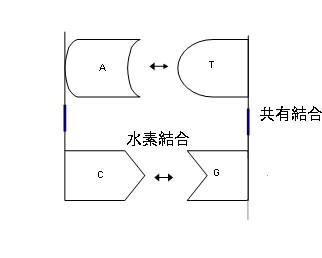
�@DNA���\������e�k�N���I�`�h�͋��L�����Ɛ��f�����Ƃ���2��ނ̌����Ō`������Ă���B
�@�e�k�N���I�`�h�ɂ����郊���_�Ƃ��ׂ̗̃k�N���I�`�h�̓������L����������1�{�����`������B �����Ă��̍���2�{���Ƃ��ɁA�e�k�N���I�`�h�̉���������f��������B����̈����t�� �����Ƃ͂��̐��f�����������B���̉�����Ƃ����̂�2�{���Ƃ��̕ʂ�DNA�����m�̉��� �ł���B�����āA���̐��f����������Ƃ��́AA��T��G��C�ƕK�����Ȃ��B��������g�\���N ���b�N�̑���Ƃ����B
�@PCR�͖ړI�ƂȂ�DNA�z��邽�߂̕��@�ł���B�܂��A����1�̕��q����n�߂āA���� DNA���q�̃R�s�[���r�I�Z���Ԃő�ʂɐ����ł���B
�ϐ����A�j�[�����O���L�����ϐ����J��Ԃ����Ƃɂ��ڕW���q�Ɠ����2�̊��S�� ��{��DNA�����������B
�@DNA���q�̒����𑪂�Ƃ��ɗ��p�����BDNA���q���}�C�i�X�ɑѓd���Ă��邽�߂� �A�d�E�ɂ������ƃv���X�ɂ������Ĉړ����錻�ۂ𗘗p���Ă���B�Q���͕��q�̂� �邢�̂悤�ɍ�p����̂ŁA�����ȕ��q�͑傫�ȕ��q�����e�ՂɃQ�������ړ�����B �����傫���̕��q�͓������x�œ����̂ň��̎��ԓ��ł͏����ȕ��q�͑傫�ȕ��q�ɔ� �ׂĂ�艓���ցi���������j�ړ����邱�ƂɂȂ�B(�}4)
�@����n�t����A�z���܂ވ�{�����q�����ׂĎ��o�������Ƃ���B���̂Ƃ��A
���̑���z����v���[�u�Ƃ�ԁi �j�B���̃v���[�u
�������Ȏ��C�r�[�Y�ɕt�������Ă����A���o�����������q�̓������n�t�ɂ���A�悭�����Ď���
�������ƁA�����܂ޔz�q���v���[�u�Ɛ��f��������B�����Ŏ��𗘗p���Ď��C�r�[�Y��e
��̕ǂɋz�����܂܁A�n�t���������Ƃɂ��v���[�u�Ɍ��������z�q���c���Ƃ������@
�ł���B(�}5)
�j�B���̃v���[�u
�������Ȏ��C�r�[�Y�ɕt�������Ă����A���o�����������q�̓������n�t�ɂ���A�悭�����Ď���
�������ƁA�����܂ޔz�q���v���[�u�Ɛ��f��������B�����Ŏ��𗘗p���Ď��C�r�[�Y��e
��̕ǂɋz�����܂܁A�n�t���������Ƃɂ��v���[�u�Ɍ��������z�q���c���Ƃ������@
�ł���B(�}5)
�@���̘_���́ADNA �R���s���[�^�Ƃ������삪�ł��邫�������ƂȂ����_���ł���B ���̘_���ł�Adleman ��NP���S���Ƃ���Ă���L���n�~���g���p�X����DNA�ɕ����� ��DNA�ł̎������s�����Ƃʼn��邱�Ƃɐ��������B�܂�AAdleman�͗L���n�~���g�� �p�X���ɑ���A���S���Y����DNA�̎����ɋA���������ƂɂȂ�B
�@Adleman �͎��g�̍l����DNA�v�Z�̗͂�NP���S��������model���g���ĉ������Ƃɂ�� �������B����NP���S���̒��őI�ꂽ�̂��A�L���n�~���g���p�X���ł���B�}1�̉E �̃O���t�̖��ł���B
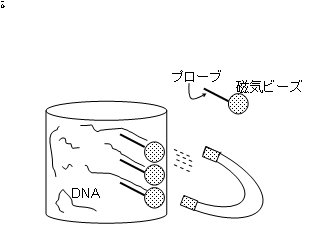
���ۂɁAAdleman ���������L���n�~���g���p�X���͐}6�̂悤��7���_,14�ӂ̗L���n�~�� �g���p�X���ł���B�܂��A���̖��͒��_�ɂ���ꂽ������ǂ��Ă����A�n�~���g�� �p�X�邱�Ƃ��ł���悤�ɁA�Ӑ}�I�ɔԍ��t�����s���Ă���B�{�_���ł́A�����̕� �X�㒸�_�̖��O��}7�̂悤�ɕύX����B
�S�Ẳ�������ݒׂ��ɒT���Ή��ɂ��ǂ蒅����������Ȃ����A�v�Z�@�Ȋw��A����ł͉��� ����܂łɍň��A�w�����Ԃ������Ă��܂����Ƃ����肦��B�i��L�̖��̓X�P�[���������� �̂ŁA�����ɉ��邱�Ƃ͉\�ł���j���̖���Adleman ��,DNA�R���s���[�^���g���āA ��������step�ނ��ƂŌ����I�Ȏ��Ԃʼn������̂ł���B���̎菇���ȉ��Ɏ����B
�@���_�ƕӂ�������B�܂��A�e���_�A�e�ӂ�������ۂ̎菇�Ƃ��ẮA�{�_���ł͕��G��DNA�z��Œ��_��������A�����āA�����ӁA���_���Ȃ��悤�ɔz���U�蕪����B�ڍׂ�[3]���Q�l�B ��Ƃ��āAstart�����Ԃ̕������ɂ��Đ�������B
�@�菇�P�Ő������ꂽ���������ꂽ�S�Ă�DNA���i�ӁA���_�S�ājDNA���K�[�[�Ƃ����y�f�� �ꏏ�ɂ��Ď����ǂɂ����B�ӂ͒��_�̃��g�\���N���b�N����ɂ���Č��߂Ă����̂ŁA ���̕ӂ�\��DNA�����_��\��DNA�̂��Ė̂悤�ɗp������B����́ADNA�̑���ɂ�� �A�j�[�����O�̐����𗘗p�������̂ł���B(�}9)
�@step�Q�Ő������ꂽ�p�X����Astart�͂��܂�goal �ŏI���p�X��\��DNA�z���S�ĕ��� ����B���̍�Ƃ́APCR�i�|�������[�[�A�������j�𗘗p����B(�}10)
�@step�R�œ���ꂽ�p�X�̒�����A����͒��_��7�̃p�X�݂̂�S�ĕ�������B ���̍�Ƃɂ́A�Q���d�C�j�����g�p�����B(�}4)
�@���̎��_��step�S�œ���ꂽ�p�X�Ƃ����̂́Astart �Ŏn�܂�Agoal�ŏI���A ����ɒ��_��7�̃p�X�݂̂��c���Ă���B���̎c�����p�X�̒�����A�S�Ă̒��_�� �ʂ����p�X�݂̂�����B�}�O�l�e�B�b�N�@�𗘗p���āA���A�����Ƃ��ׂ� �̒��_�ɑ��Č������s���B(�}11)
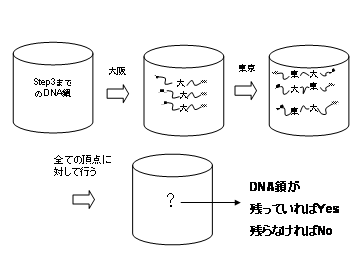
�@�Ō�ɁAstep�T���I������i�K�ŁADNA���c���Ă��邩�ǂ������m�F����B����DNA���c���Ă���Ȃ�A���̎c���Ă���DNA�Ƃ����̂��A���̉���\�����̂ƂȂ�B
�@�ȏオAdleman�̎����ł���B
�@Adleman �̗L���n�~���g���p�X���̉�@�͒P�ɂЂƂ̎����ɗ��܂炸�A���Ɉ�ʓI�� �v�Z���J�j�Y��������Ƃ�����B���̂��Ƃ���A���̉��Z�p���_�C����DNA�v�Z�ɂ��� ��"Adleman�p���_�C��"�ƌĂ��
�@���̘_���́AAdleman�̘_����A����ȍ~�ɏo��DNA Computer�̘_���Ɠ����悤�ɁA�^���� �ꂽ����\�����钆�S�̃��J�j�Y���Ƃ��āADNA�����g�p���Ă���T�O���f���ł���B�܂� �A�㔼�ɂ͂��̃��f���𗘗p���Ď����ʼn��Z���s�����߂̎������s���A�C�f�A���Љ��Ă� ��B�^����ꂽ����2�i���ŏ������邽�߂̍H�v���Ȃ���Ă���B���̘_�����o��ȑO��DNA �R���s���[�^�ɔ�ׂāA���̃��f���̗��_�́ADNA���̊g����v�����Ȃ�RAM(ramdom access memory)�������Ă��邱�ƁA�y�f��K�v�Ƃ��Ȃ����ƁA�ޗ����ė��p�ł��邱�Ƃł���B�܂� �A�ڕW�Ƃ���4�������Ă���B��ڂ�DNA�R���s���[�^ �ɂ���āA�ėp�ړI�̃A���S�� �Y�����\�z����B���̔ėp�ړI�̃A���S���Y���Ƃ͒T�����̍L���N���X����������A���S�� �Y���ł���B�L���N���X�Ƃ́ANP���S��肩�A����ȏ�̂��̂ł���B��ڂ͂قǂقǂ̗� ��DNA��NP���S��肩����ȏ�̖��������邱�Ƃ��ؖ����邱�ƁB�O�ڂ͍\���⋤�L���� �̔j���DNA Computer �ɂ͖{���I�ł͂Ȃ����Ƃ������B�l�ڂ�Sequence-specific separat ion �͔ėp�ړI�̕��q�R���s���[�^���\�z����ɂ́ADNA�H�w�Ƃ��Ă��\���ł��邱�Ƃ����� �B�ȏ�4���ڕW�Ƃ��Ă������Ă���B
�@���̕����͂ЂƂ�bit����\�����邽�߂ɁA�P���DNA���Ƃ���DNA���Ƒ���I�ȊW ��DNA���̏��Ђ����f�����ł��������Ƃɂ��\������B�܂��A���̃��f���𗝉����邽 �߂Ɋ�{�I�Ȍ��̐���������B
����Ƃ���(7,3)library�Ƃ́A{0000000,0010000,0100000,1000000,0110000,1010000,1100000,1110000}�̂��Ƃł���B �@���ɁA���̕\�����@���������B �}12��sticker model�̏��\�����@�̊T���}�ł���
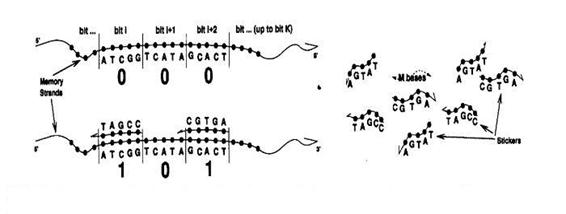
�@memory strand �̒�����N����Abit�̈�͊e�XK���݂��A�e�X��bit�̈�̒�����M�Ƃ���B
���̂Ƃ��A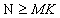 �ł���B���ꂼ��memory strand�ɂ�N�̈�ȊO�ɔėp�̈�Ȃ�̈�������Ă���B
�܂��A�����Ƃ̓k�N���I�`�h�̗�̂��Ƃł���B�Ⴆ�AAGCTT�ł���Β���5�ł���Bmemory
strand�ɂ͐擪�ɓ���̔z��������Ă�����̂����݂���B���̔z���initial set�ƌĂԁB
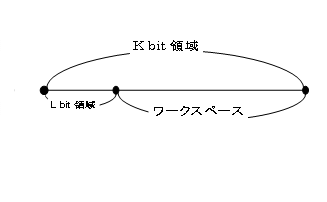
����initial set������tube�̂��Ƃ�mother tube�ƌĂԁBinitial bit ��K bit����L bit�̖�
��ɂ��^����ꂽ���͂������邽�߂̗̈�̂��Ƃł���B�܂��A(K-L)bit�͏��߂̏��
�Ƃ��đS��bit ��off�ɂ��Ă���B����(K-L)bit�̈�̂��Ƃ����[�N�X�y�[�X�ƌĂсA����
�v�Z����Ȃ��ʼn��Z���s����X�y�[�X�Ƃ��ė��p�����B
�ł���B���ꂼ��memory strand�ɂ�N�̈�ȊO�ɔėp�̈�Ȃ�̈�������Ă���B
�܂��A�����Ƃ̓k�N���I�`�h�̗�̂��Ƃł���B�Ⴆ�AAGCTT�ł���Β���5�ł���Bmemory
strand�ɂ͐擪�ɓ���̔z��������Ă�����̂����݂���B���̔z���initial set�ƌĂԁB
����initial set������tube�̂��Ƃ�mother tube�ƌĂԁBinitial bit ��K bit����L bit�̖�
��ɂ��^����ꂽ���͂������邽�߂̗̈�̂��Ƃł���B�܂��A(K-L)bit�͏��߂̏��
�Ƃ��đS��bit ��off�ɂ��Ă���B����(K-L)bit�̈�̂��Ƃ����[�N�X�y�[�X�ƌĂсA����
�v�Z����Ȃ��ʼn��Z���s����X�y�[�X�Ƃ��ė��p�����B
�@stickers��memory strand �̑Ή�����bit�̈�ɂ͂�����ƂŁA2�i����\������B bit 1 �ɂ�S1�Abit 2 �ɂ�S2�Abit k �ɂ�Sk�Ƃ����悤��stickers�͊e�X���߂�ꂽbit �ɂ����ɂ͂�������������Ă���B(S�Ƃ�sticker��\��) stickers��memory strand �̏����bit�̈�ɂ͂�������A���̂͂����bit�̈��"1"�Ƃ���B�͂���Ȃ��� ���"0"�Ƃ���
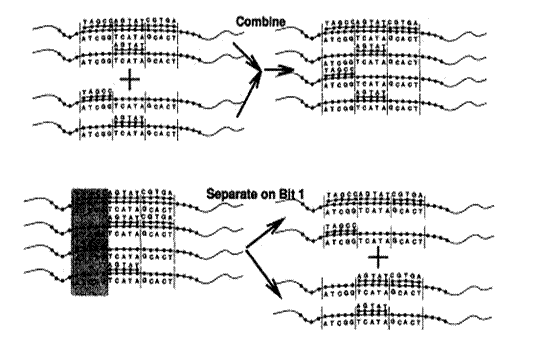
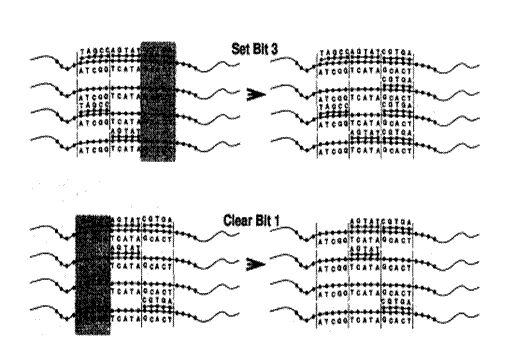
�@���̕����ɂ́Atube �ɑ��āA4�̊�{���Z���p�ӂ���Ă���Bcombine, separate on Bit X, Set Bit Y, Clear Bit Z �ł���B(�}14)
�@���܂Œ�`�������f���̗͂��ؖ����邽�߂ɁANP���S���ȏ�̓���Ƃ���Ă������ �ŏ��핢�����������Ƃɂ���B�ŏ��핢���Ƃ͑S�̏W��U���W��Ci�ii={1�cB}�j�̘a�W ���Ŕ핢����ꍇ�A�ǂ̑g�ݍ��킹���S�̏W��U�̗v�f��핢���邽�߂ɍŏ��̘a�W���Ŕ핢�� �邱�Ƃ��ł��邩��₤���ł���B
U�F�S�̏W��
C= {C1, �c,CB} , Ci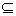 {1,�c,A}, �ŏ������W��I
{1,�c,A}, �ŏ������W��I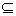 {1,�c,B} , ��i
{1,�c,B} , ��i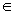 ICi = {1,�c, A}
�A���AA,B�͎��R���Ƃ���B
ICi = {1,�c, A}
�A���AA,B�͎��R���Ƃ���B
�@���̗�Ƃ��� U={1,�c,10} ,C={{1,3,5,7,9}=C1, {2,4,6,8,10}=C2, {3,6,9}=C3, {1,2,3,5,7}=C4�Ƃ���B U��핢������̂Ƃ��āAC1��C2=U �ƁAC2��C3��C4=U �ȂǑ����l�����邪�ŏ��핢�Ȃ̂ŁAI={1,2} �ƂȂ�B���̖�����{���Z�𗘗p���ĉ������Ƃɂ���
�ȏ�̃A���S���Y���ɏ]���A���������Ă݂�B
�@�}15���̖��̕������Ə�������\���Ă���Bmother tube�擪��B bit ����0,1�̑S�� �̑g�ݍ��킹����mother tube��p�ӂ���B�܂��AA bit �͂��ׂĂ�bit �̈��0�ɂ��Ă��� �B
�@mother tube �̐擪��bit ��1�ƂȂ��Ă�����̂ƁA0�ɂȂ��Ă�����̂���{���Zseparate�ɂ�蕪������B�����āA�擪��bit �̈悪1�ɂȂ��Ă���tube�̓����Ă���e ���stickers �𓊓�����B�����āA����������tube��������x�A��{���Zcombine�� ��荇�킹��B���̍�Ƃ���̓��͂�\��bit �̈�i���ł�B bit�j�̑S�Ăōs�� �B(�}16)���̍�Ƃ��I���邱�ƂŁA(K,L)library�������B
�@�ȏ�̍�Ƃ���ɂ����čs���ƁA�\1��(K,B)library��B �\1�ɂ��Đ�������B1�s��1��memory complex��\�����Ă���B �擪4���B bit ���W���A�܂�C= {C1, �c,CB}�B�c���10���C= {C1, �c,CB}�Ɋ܂܂� ��v�f��\���Ă���B�܂��A�S�Ă̗v�f��핢����mother tube�ɂ�1��ڂɁ����A�v�f��1�ł� �핢����Ă��Ȃ����̂ɂ́~���L����Ă���B
�@�Ⴆ�A�\�ォ��3�s�ڂ̏ꍇ�A 00011110101000��C4={1,2,3,5,7}��\�����Ă���B �܂��A12�s�ڂ̏ꍇ�A 11001111111111��C1��C2={1,2,3,4,5,6,7,8,9,10}��\�����Ă���B�܂��A�S�Ă̗v�f��핢���Ă���̂�1��ڂɂ́����L�ڂ���Ă���B ������́A������₷�����邽�߂ɕ\��p�������A���ۂ�DNA�����ł͕\�������킯�ł͂Ȃ��B
�@
| memory complex�� | C1 | C2 | C3 | C4 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| �~�@ | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| �~�@ | 0 | 0 | 0 | 1 | 1 | 1 | 1 | 0 | 1 | 0 | 1 | 0 | 0 | 0 |
| �~�@ | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 1 | 0 |
| �~�@ | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | 1 | 0 | 1 | 0 | 1 |
| �~�@ | 1 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | 1 | 0 | 1 | 0 | 1 | 0 |
| �~�@ | 0 | 0 | 1 | 1 | 1 | 1 | 1 | 0 | 1 | 1 | 1 | 0 | 1 | 0 |
| �~�@ | 0 | 1 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 0 | 1 |
| �~�@ | 0 | 1 | 1 | 0 | 1 | 1 | 1 | 1 | 0 | 1 | 0 | 1 | 1 | 1 |
| �~�@ | 1 | 0 | 0 | 1 | 1 | 1 | 1 | 0 | 1 | 0 | 1 | 0 | 1 | 0 |
| �~�@ | 1 | 0 | 1 | 0 | 1 | 0 | 1 | 0 | 1 | 1 | 1 | 0 | 1 | 0 |
| ���@ | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
| ���@ | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
| �~�@ | 1 | 0 | 1 | 1 | 1 | 1 | 1 | 0 | 1 | 1 | 1 | 0 | 1 | 0 |
| ���@ | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
| ���@ | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
| ���@ | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
�\1.(K,B)library
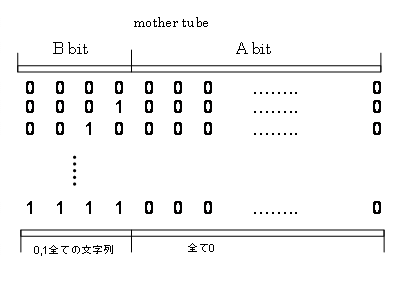
�@�ȏオ�Astickers model�ōŏ��핢���������ۂ̃A���S���Y���ł���B �@��������́Astickers model �����ۂ�DNA�Ŏ�������ۂ̕��@�A���ӓ_���Љ��B
�@���̎����́A2��tube���Ăѐ��a�����邱�Ƃɂ���Ď��s�\�ł���B�������A���̒P�� �ȕ��@�́A��������A�s�y�b�g��tube�����o�����肷��Ƃ��ɁA�אS�̒��ӂ��K�v�ł���B �����A�s�y�b�g�ɂ����킸���ȗʂ�DNA���c���Ă����Ƃ��Ă��A����͑�ςȌ덷�̌����ɂ� ��B�Ȃ��Ȃ�A���̉��Z�ɂ����āA���͂�\������DNA���A�W�̂��镪�q���͂��߂��班 �Ȃ���Ԃł��邩��ł���B
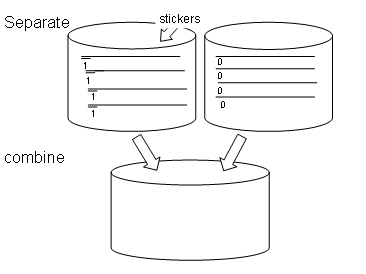
�@���̎����́Asticker model�ň�ԏd�v�ł���B�Ȃ��Ȃ�A����\������DNA�����Ă� �����ŁA����g�p����鉉�Z�ł��邩��ł���B�����̒��ӓ_��memory complex ���m�� �����鎞�ɁA����memory complex�ɃA�j�[������Ă���sticker ��sticker ���A�j�[������ �Ă���bit�̈�ɂ͂������ԂłȂ�������Ȃ��B�܂�A����bit�̈悩��Asticker�� �͂���Ă͂����Ȃ��̂ł���B�Ή���Ƃ���separation �̎����ɂ́A�v���[�u���g���B�v�� �[�u�͑Ή�����bit�̈�ɂ��ꂼ��p�ӂ��Ă����B���}�O�l�e�B�b�N�r�[�Y�@�Q��
�@�v���[�u�́A�Ή�����bit�̈�Ƒ���W�̂���DNA�ō\������Ă��邪�Asticker���͂��� �Ȃ��悤�ɂ��邽�߂ɁAsticker ���A�j�[������Ă���͂����ア�͂�memory complex�� �Ƃ炦�Ȃ��Ă͂����Ȃ��B(�}17)
�@setting �͑ΏۂɂȂ�bit�̈�ɒ���sticker���A�j�[������Bset ���s��bit�̈�ɑΉ���
��sticker���ʂɗp�ӂ��Ă����B�����āAset���s��bit�̈���܂�memory strand�Ƃ���
��ʂɗp�ӂ���sticker������������B��ʂɗp�ӂ������߁A�A�j�[������Ȃ�����sticker
�͎�菜���B
�@clearing �͌��i�K�ł͎����\�łȂ��Ƃ���Ă��邪�Aclearing�̎������ł��Ȃ���
��sticker model �̌v�Z�\�͕͂ς��Ȃ��Ƃ���Ă���B
�A���S���Y���Ƃ���
�ƁA�Љ�����A���S���Y���̇A(K,L)library��p�ӂ��邱�Ƃ�sticker��1�cL��bit�̈� ���ŁAmemory strand�Ƀ����_����sticker���������Ȃ���Ȃ�Ȃ��B���̎菇���ȉ��Ɏ� ���B
�@memory strand��2�̓������ʂɕ�����B�ЂƂɂ́A1�cL�̑S�Ă�bit���������ʂ� sticker ��������B����ŁA1�cL�̑S�Ă�bit�̈��sticker ���A�j�[������邱�ƂɂȂ�B ���̂Ƃ��A�A�j�[������Ȃ�����sticker �͎�菜���A���̌�A�ŏ��ɂ킯�����̂ƌ����� ���A�܂��A�M����A�����Ă����P�x�Asticker ���𗣂�����B���̌�A����1�x�A��₷���� ��sticker ���A�j�[��������B
�@���̕��@�Ŏ��ۂ�(K,L)library������Ă��Ƃ��A�ǂ̂��炢�̊m����(K,L)library��
�ł��邩�Ƃ������Ƃ��l����B��̎菇�Ŋebit �̈�̓A�j�[���������̂Ƃ���Ȃ�
���̂�1/2�̊m���������ƂɂȂ�B�܂��Abit�̈�͑S�č��킷�� L bit�ɂȂ�̂ŁA
L���̓����bit �X�g�����O�������m����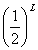 �ƂȂ�B�܂��A
L���̓����bit �X�g�����O������Ȃ��m����1-
�ƂȂ�B�܂��A
L���̓����bit �X�g�����O������Ȃ��m����1-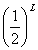 �ƂȂ�B
(K,L)library�œ����bit�X�g�����O������Ȃ��m����2
�ƂȂ�B
(K,L)library�œ����bit�X�g�����O������Ȃ��m����2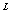 ��bit �p�^�[�������݂���̂ŁA
��bit �p�^�[�������݂���̂ŁA
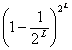 �ƂȂ�B������v�Z����ƁA�e�X��memory complex�͏��Ȃ��Ƃ�1��ł��悻63%�̉\����
�����č����B���̃p�[�Z���e�[�W��memory strand �� 2 �ȏ���������邱�Ƃɂ����
���炩�ɑ��₷���Ƃ��\�ł���B���̎�i�͉��w�ʘ_�I�ɂ̓G���[�ɋ����p�[�Z���e�[�W
�Ƃ�����B
�ƂȂ�B������v�Z����ƁA�e�X��memory complex�͏��Ȃ��Ƃ�1��ł��悻63%�̉\����
�����č����B���̃p�[�Z���e�[�W��memory strand �� 2 �ȏ���������邱�Ƃɂ����
���炩�ɑ��₷���Ƃ��\�ł���B���̎�i�͉��w�ʘ_�I�ɂ̓G���[�ɋ����p�[�Z���e�[�W
�Ƃ�����B
�@�ŏI�I�ɉ��邽�߂ɂ͗n�t�̒���memory complex�����݂��邩�A���Ȃ��������o�ł��� �K�v������B�����A�������݂���Ȃ�A���Ȃ��Ƃ��ЂƂ�memory complex��P���ł��� �K�v�����邵�A�����āA����ɃA�j�[������Ă���sticker���m�F����K�v������B
�@memory complex�̌��o�́A�e�X��memory strand �Ɍu�����̃��x�����O���{�����Ƃɂ�� �\�ƂȂ�B�܂��A�A�v���[�`�Ƃ��ẮA�S�Ă̓����Ƃ݂Ȃ����memory complex���܂�� ����n�t�̒�������𗘗p���ē����ƂȂ�DNA�z������ۂɖڂŌ��Ċm�F����̂ł���B
�@stickers model����̓I�ȃV�X�e���Ƃ��ďЉ��B
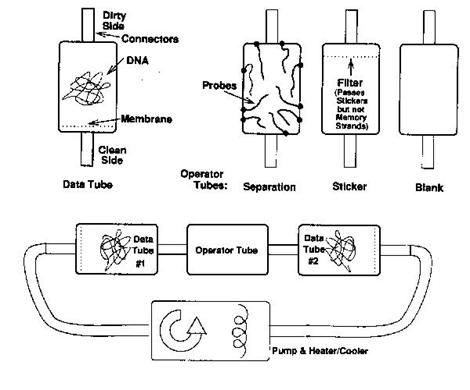
�@�}18��stickers model���V�X�e��������stickers machine�ł���B���̑��u��2��ނ�Tube �ƌĂ��ǂɂ��\������Ă���B�ЂƂ́A����\������S�Ă�DNA�������Ă���Data Tube �Ɗe��{���Z���s��operator Tube �ł���BData Tube �ɂ�memory strand �� stickers ���ʂ��Ȃ�Membrane(����)�����݂��A���̔����͏���\������DNA �ɋɐ���^����B������ �߂��R�l�N�^�[���̐��clean side �ƌĂсAmemory strand ,memory complex, stickers, tube���܂�DNA�͑��݂����A�␅�A�M���݂̂����݂���B���̔��̃R�l�N�^�[���� dirty side �ƌĂсAmemory strand ,memory complex, stickers, tube���A����\������ DNA�����݂���BOperator Tube��separation �̂��߂̃v���[�u���Œ肵��Separation Tube�ƁA stickers�͒ʉ߂ł��邪memory strand �͒ʉ߂ł��Ȃ��t�B���^�[��������sticker Tube, �P�Ɋǂ��Ȃ������̖���������������blank Tube ��3��ނ�����B
�@���̑��u�͐}18���i�̂悤�ɁA������Data Tube ��u���A���̊Ԃɂ���operator Tube���R ���s���[�^����̃��{�b�g�̂悤�Ȃ��̂ŕK�v�ɉ����Ēu�������A�����DNA �����̃A���S ���Y���i��ɃA�j�[�����O�Ȃǁj�ɍ��킹�č��E���玩�R�ɉ�����������₽�������𗬂� ��悤�ɂ������̂ŁA��Ɏ�����4��ނ̉��Z�������ōs����B���̂��߁A�ŏ��ɖ��̐ݒ� �����邾���ŁA�����ʼn��Z���s�����Ƃ��\�ł���B
�@�ȏ�Astickers model���Љ���B����model ��NP���S���ȏ�̖��������͂��������킹 �Ă���A�܂��A�y�f������Ȃ����ƁAram�̋@�\���ʂ����Ă���Ƃ������_�������Ă���B �܂��A���̐ݒ���s�������Ŏ����ʼn��Z���s�����u�̏Љ������Ă���B���邽�߂�4�� �̊�{���Z�A����separate��DNA�H�w�ɂ����Ă�DNA�R���s���[�^���\�z���邤���ŏ\���ł��� ���Ƃ�������Ă���B�܂��AAdleman�̎����ɔ�ׁA���肠��ʂ�DNA�Ōv�Z���s�����Ƃ��\ �ł���B
�@�{�_����DNA �R���s���[�^�̘_���Ƃ��āAAdleman��Molecular computation of solution to combinatorial problems��Sam Roweis�AW.K.Rothemund���A Sticker-Based Model for DNA Computation���Љ���BAdleman�̘_���͉����Ȃ��Ƃ��납��A����DNA�R���s���[�^ �Ƃ���������N���������炵���_���ł���B�܂��ADNA���g�����v�Z�X�^�C���ł���܂Œʏ� �̌v�Z�@�ł͉����̂�����Ƃ���Ă���NP���S�����������̂ŁA�v�Z�@�Ȋw�Ƃ��Ă��V�� �ȃA���S���Y���̊J���ɂ��Ȃ����B
�@stickers model �̘_���́ANP���S���������\�͂̂��鉉�Z��p�ӂ��A���̉��Z�𗘗p���� NP���S���ł���ŏ��핢�����������B�����āARAM�̋@�\�������Ă���A�y�f���g��Ȃ��� �悢�Ƃ������_���������킹�Ă���B�܂��A�����̃A�C�f�A�₻�̎����ɂ�����DNA�����̉\ ���ɂ��Ă��Љ��Ă���B
�@���ꂩ��̎��̕��j�Ƃ��āADNA �R���s���[�^�̊W���镪��ɓ���Љ�I�ɗǂ��e����ǂ� �z�ނ��Ƃ̂ł��錤����J�����s���Ă��������Ǝv���B
�@DNA �R���s���[�^�ɂ��Ă��w�����������܂��������H�Ƒ�w��w�@�@�؉���y�����A�R����K�����A�@ ������w��w�@�@�L�c ���K�y�����Ɋ��ӂ������܂��B
�@�Ō�ɁA�����҂Ƃ��Ă͂Ȃ�ł��邩�A�܂��A�_����ǂނɂ������Ă̗l�X�Ȓ��ӁADNA �R���s���[�^�𑲘_�̃e�[�}�Ƃ��Ĉ����Ă�����������{���u�y�����Ɋ��ӂ������܂��B